Tensorflow Probability 기본 (1)
( 참고 : coursera : Probabilistic Deep Learning with Tensorflow2, Tensorflow official website )
Contents
- Distribution
- Univariate ( Normal, Bernouli )
- Multivariate
- Covariance
- Full covariance matrix
- Cholesky Decomposition
- Independent
- Trainable Distribution
import tensorflow as tf
import tensorflow_probability as tfp
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
tf.random.set_seed(123)
tfd = tfp.distributions
1. Distribution
1-1. Univariate
(1) Normal Distn
- loc : 평균 & scale : 표준편차
normal = tfd.Normal(loc=0.,scale=1.)
normal.sample(5)
normal.sample((3,4)) # or normal.sample([3,4])
normal.prob(0.5)
normal.prob([0.5,0.4])
normal.log_prob(0.5)
normal.log_prob([0.5,0.4])
(2) Bernoulli Distn
bern = tfd.Bernoulli(probs=0.7)
bern2 = tfd.Bernoulli(logits=0.847)
-
bern : \(p=0.7\)
-
bern2 : \(\text{log}(\frac{p}{1-p}) = 0.847\)
1-2. Multivariate
쉬운 이해
- event shape는 “차원”
- batch shape는 “분포의 개수”
(1) Multivariate Normal Distn
mvn = tfd.MultivariateNormalDiag(loc=[-1.,0.5],
scale_diag=[1.,1.5])
batched_normal = tfd.Normal(loc=[-1.,0.5],
scale=[1.,1.5])
- mvn : Multivariate 1개 ( : event shape = 2 )
- batched_normal = Univariate 2개 ( : batch_shape = 2)
mvn.log_prob([0.1,0.2])
# mvn.log_prob([0.1,0.2,0.3]) -> 에러 ( 2차원에 맞게끔 해야 )
(2) BATCHED Multivariate Normal Distn
batched_mvn = tfd.MultivariateNormalDiag(
loc=[[-1.,0.5],[2.,0.],[-0.5,1.5]],
scale_diag=[[1.,1.5],[2.,0.5],[1.,1.]])
- batch_shape = 3
- event_shape = 2
2. Covariance Matrix
2-1. Full Covariance Matrix
Spherical ( Isotropic ) Gaussian이란?
- 각 component의 variance가 동일한 경우! & ( random vector being independent )
- ex) \(\Sigma = \sigma^2 I\)
Full covariance matrix
- Isotropic과는 다르게, correlation이 존재! ( dependent )
tfd.MultivariateNormalTriL를 사용해서 full covariance Gaussian 지정 가능
tfd.MultivariateNormalTriL.
loc: \(\mu\)scale_trila lower-triangular matrix \(L\) such that \(LL^T = \Sigma\).
\(d\)-dim r.v에서, lower-triangular matrix \(L\) 는 다음과 같다.
\[\begin{equation} L = \begin{bmatrix} l_{1, 1} & 0 & 0 & \cdots & 0 \\ l_{2, 1} & l_{2, 2} & 0 & \cdots & 0 \\ l_{3, 1} & l_{3, 2} & l_{3, 3} & \cdots & 0 \\ \vdots & \vdots & \vdots & \ddots & \vdots \\ l_{d, 1} & l_{d, 2} & l_{d, 3} & \cdots & l_{d, d} \end{bmatrix}, \end{equation}\]# (1) Mean
mu = [0., 0.]
# (2) Covariance
scale_tril = [[1., 0.],
[0.6, 0.8]]
sigma = tf.matmul(tf.constant(scale_tril), tf.transpose(tf.constant(scale_tril)))
#-------------------------------------------------
mvn = tfd.MultivariateNormalTriL(loc=mu, scale_tril=scale_tril)
scale_tril에는, \(\Sigma\)가 들어오는 것이 아니라, \(L\)이 들어와야한다
2-2. Cholesky Decomposition
covariance matrix : \(\Sigma = LL^T\). 그렇다면, 왜 \(\Sigma\) 대신 lower-triangular matrix인 \(L\)을 사용해서 표현할까?
Covariance matrix (\(\Sigma\))는 다음의 특성을 따르기 때문이다!
-
It is symmetric
-
It is positive (semi-)definite
NB: A symmetric matrix \(M \in \mathbb{R}^{d\times d}\) is positive semi-definite if it satisfies \(b^TMb \ge 0\) for all nonzero \(b\in\mathbb{R}^d\).
If, in addition, we have \(b^TMb = 0 \Rightarrow b=0\) then \(M\) is positive definite.
Cholesky Decomposition는, 위의 특징을 사용하여, symmetric positive-definite matrix \(M\)를 \(LL^T = M\)처럼 분해하는 것을 의미한다. 따라서 우리는 위 2-1에서 말한 것 처럼, covariance matrix를 나타낼 때 tfd.MultivariateNormalTriL.을 사용할 수 있는 것이다.
# (1) Mean
mu = [1., 2., 3.]
# (2) Covariance
sigma = [[0.5, 0.1, 0.1],
[0.1, 1., 0.6],
[0.1, 0.6, 2.]]
scale_tril = tf.linalg.cholesky(sigma)
#-------------------------------------------------
mvn = tfd.MultivariateNormalTriL(loc=mu, scale_tril=scale_tril)
3. Independent
tfd.Independent( ,reinterpreted_batch_ndims= )
independent + batch distribution = multivariate distribution ( batch size가 event size로 흡수 됨 )
여러 개의 univariate distn을, (correlation이 서로 0인) multivariate distn으로 묶어줄 수 있다.
# (1) Multivariate distn
mvn = tfd.MultivariateNormalDiag(loc=[-1.,0.5],
scale_diag=[1.,1.5])
# (2) Batch distn
batched_normal = tfd.Normal(loc=[-1.,0.5],
scale=[1.,1.5])
reinterpreted_batch_ndims : controls the number of batch dims which are absorbed as event dims
-
reinterpreted_batch_ndims=1 : 변환 이후의 event_shape는 [a]
reinterpreted_batch_ndims=2 : 변환 이후의 event_shape는 [a,b]
normal_2d_ind = tfd.Independent(batched_normal,reinterpreted_batch_ndims=1)
결과
batched_normal의 shape : (batch shape=[2], event shape=[])normal_2d_ind의 shape : (batch shape=[0], event shape=[2])
normal_2d_ind.log_prob([0.2,0.4]))
mvn.log_prob([0.2,0.4])
- 위의 두 값은 같다. 왜냐하면, mvn의 경우에 서로 correlation이 없는 것으로 지정을 해줬기 때문이다.
4. Trainable Distribution
4-1. Intro & 분포 정의하기
Distribution의 parameter에 constant대신 tf.Variable(xxx)를 넣어주면, 이는 학습가능한 분포로써 정의된다.
Assumption
- 실제 정답 분포 : rate = 0.3
- 학습하고자 하는 분포의 initial state : rate = 1.0
# (1) 일반 distn
exponential = tfd.Exponential(rate=0.3,name='exp')
# (2) Trainable Distribution
exp_train = tfd.Exponential(rate=tf.Variable(1.,name='rate'),name='exp_train')
exp_train.trainable_variables
4-2. Loss Function 정의
방금 생성한 Exponential 분포를 학습시켜보겠다.
그러기 위해 우선 loss function을 정의해야하는데, 확률에서 많이 사용하는 nll(negative log likelihood)를 사용하겠다.
def nll(x_train,dist):
return -tf.reduce_mean(dist.log_prob(x_train))
Loss를 계산하고, 이의 기울기를 구하는 함수 get_loss_and_grad
@tf.function
def get_loss_and_grad(x_train,dist):
with tf.GradientTape() as tape:
tape.watch(dist.trainable_variables)
loss = nll(x_train,dist)
grads = tape.gradient(loss,dist.trainable_variables)
return loss,grads
4-3. Train 함수
train_loss에 epoch별로의 train loss를 담는다train_rates에 epoch별로의 변화하고 있는 parameter ( rate )를 담는다
def exp_dist_optimize(data,dist,n_epoch=15):
train_loss = []
train_rate = []
opt = tf.keras.optimizers.SGD(learning_rate=0.05)
for epoch in range(1,n_epoch+1):
loss, grads = get_loss_and_grad(data,dist)
opt.apply_gradients(zip(grads,dist.trainable_variables))
rate=dist.rate.value()
train_loss.append(loss)
train_rate.append(rate)
print('Epoch {:03d}:Loss :{:.3f}:Rate:{:.3f}'.format(epoch,loss,rate))
return train_loss,train_rate
Train data를 임의로 생성한다. (3000개)
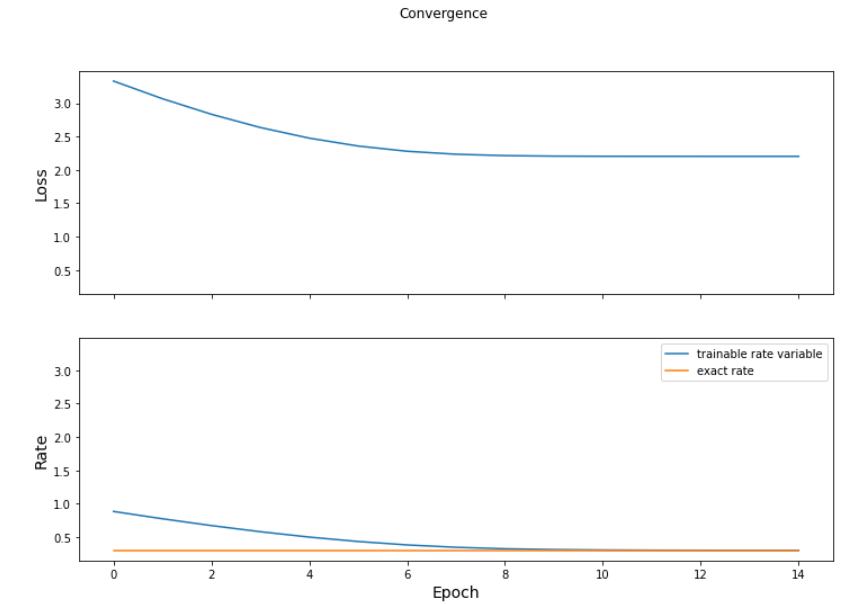
아래의 코드를 시행하면, rate=1에서 시작했던 초기값이, 학습이 진행됨에 따라 점차 0.3에 가까워진다.
sampled_data = exponential.sample(3000)
train_loss,train_rate = exp_dist_optimize(data=sampled_data,
dist=exp_train)
Epoch 001:Loss :3.331:Rate:0.883
Epoch 002:Loss :3.067:Rate:0.773
Epoch 003:Loss :2.833:Rate:0.672
Epoch 004:Loss :2.635:Rate:0.579
Epoch 005:Loss :2.476:Rate:0.499
Epoch 006:Loss :2.358:Rate:0.433
Epoch 007:Loss :2.279:Rate:0.382
Epoch 008:Loss :2.235:Rate:0.346
Epoch 009:Loss :2.214:Rate:0.324
Epoch 010:Loss :2.206:Rate:0.312
Epoch 011:Loss :2.204:Rate:0.306
Epoch 012:Loss :2.203:Rate:0.303
Epoch 013:Loss :2.203:Rate:0.301
Epoch 014:Loss :2.203:Rate:0.301
Epoch 015:Loss :2.203:Rate:0.300
4-4. 결과 비교 및 Visualization
결과 비교
- 실제 rate : 0.3
- 예측 rate : 0.3004281
pred_rate = exp_train.rate.numpy()
actual_rate = exponential.rate.numpy()
print(pred_rate,actual_rate)
0.3004281 0.3
exact_value = tf.constant(actual_rate,shape=[len(train_rate)])
fig,axes = plt.subplots(2,sharex=True,sharey=True,figsize=(12,8))
fig.suptitle('Convergence')
axes[0].set_ylabel('Loss',fontsize=14)
axes[0].plot(train_loss)
axes[1].set_xlabel('Epoch',fontsize=14)
axes[1].set_ylabel('Rate',fontsize=14)
axes[1].plot(train_rate,label='trainable rate variable')
axes[1].plot(exact_value,label='exact rate')
axes[1].legend()
plt.show()