( reference : Machine Learning Data Lifecycle in Production )
Assignment 1. TensorFlow Data Validation (TFDV)
Goal
- generate & visualize statistics from df
- infer data schema
- detect & fix anomalies
1. Setup & Imports
import os
import pandas as pd
import tensorflow as tf
import tempfile, urllib, zipfile
import tensorflow_data_validation as tfdv
from tensorflow.python.lib.io import file_io
from tensorflow_data_validation.utils import slicing_util
from tensorflow_metadata.proto.v0.statistics_pb2 import DatasetFeatureStatisticsList, DatasetFeatureStatistics
tf.get_logger().setLevel('ERROR')
2. Import Dataset
# replace '?' with NA
df = pd.read_csv('dataset_diabetes/diabetic_data.csv', header=0, na_values = '?')
df.shape # (101766, 50)
(1) Data Split
Data Split
- train : eval : serving = 70 : 15 : 15
Drop label column
- only in SERVING df
def prepare_data_splits_from_dataframe(df):
# (1) Data Split Length
train_len = int(len(df) * 0.7)
eval_serv_len = len(df) - train_len
eval_len = eval_serv_len // 2
serv_len = eval_serv_len - eval_len
# (2) Data Split
train_df = df.iloc[:train_len].sample(frac=1, random_state=48).reset_index(drop=True)
eval_df = df.iloc[train_len: train_len + eval_len].sample(frac=1, random_state=48).reset_index(drop=True)
serving_df = df.iloc[train_len + eval_len: train_len + eval_len + serv_len].sample(frac=1, random_state=48).reset_index(drop=True)
# (3) Drop label column
serving_df = serving_df.drop(['readmitted'], axis=1)
return train_df, eval_df, serving_df
train_df, eval_df, serving_df = prepare_data_splits_from_dataframe(df)
- train_df : (71236, 50)
- eval_df : (15265, 50)
- serving_df : (15265, 49)
3. Generate & Visualize Statistics
make descriptive statistics from dataset ( = EDA )
(1) Remove Irrelevant Features
- class :
tfdv.StatsOptions- params :
feature_allowlist: features to include
- params :
- 일부 변수 제거
features_to_remove = {'encounter_id', 'patient_nbr'}
approved_cols = [col for col in df.columns if (col not in features_to_remove)]
- StatsOptions class & 반드시 포함시킬 변수 지정
stats_options = tfdv.StatsOptions(feature_allowlist=approved_cols)
print(stats_options.feature_allowlist)
['race', 'gender', 'age', 'weight', 'admission_type_id', 'discharge_disposition_id', 'admission_source_id', 'time_in_hospital', 'payer_code', 'medical_specialty', 'num_lab_procedures', 'num_procedures', 'num_medications', 'number_outpatient', 'number_emergency', 'number_inpatient', 'diag_1', 'diag_2', 'diag_3', 'number_diagnoses', 'max_glu_serum', 'A1Cresult', 'metformin', 'repaglinide', 'nateglinide', 'chlorpropamide', 'glimepiride', 'acetohexamide', 'glipizide', 'glyburide', 'tolbutamide', 'pioglitazone', 'rosiglitazone', 'acarbose', 'miglitol', 'troglitazone', 'tolazamide', 'examide', 'citoglipton', 'insulin', 'glyburide-metformin', 'glipizide-metformin', 'glimepiride-pioglitazone', 'metformin-rosiglitazone', 'metformin-pioglitazone', 'change', 'diabetesMed', 'readmitted']
(2) Generate Training Statistics
output 형태 : DatasetFeatureStatisticsList
train_stats = tfdv.generate_statistics_from_dataframe(train_df, stats_options)
datasets {
num_examples: 71236
features {
type: STRING
string_stats {
common_stats {
num_non_missing: 69868
num_missing: 1368
min_num_values: 1
max_num_values: 1
avg_num_values: 1.0
num_values_histogram {
buckets {
low_value: 1.0
high_value: 1.0
sample_count: 6986.8
}
buckets {
low_value: 1.0
high_value: 1.0
sample_count: 6986.8
}
buckets {
low_value: 1.0
high_value: 1.0
...
path {
step: "readmitted"
}
}
}
- feature 개수 ( 48개 )
len(train_stats.datasets[0].features)
- data 개수 ( 71236개 )
train_stats.datasets[0].num_examples
- feature 명 ( 첫 번째 & 마지막)
train_stats.datasets[0].features[0].path.step[0] # race
train_stats.datasets[0].features[-1].path.step[0] # readmitted
(3) Visualize Training Statistics
tfdv.visualize_statistics(train_stats)
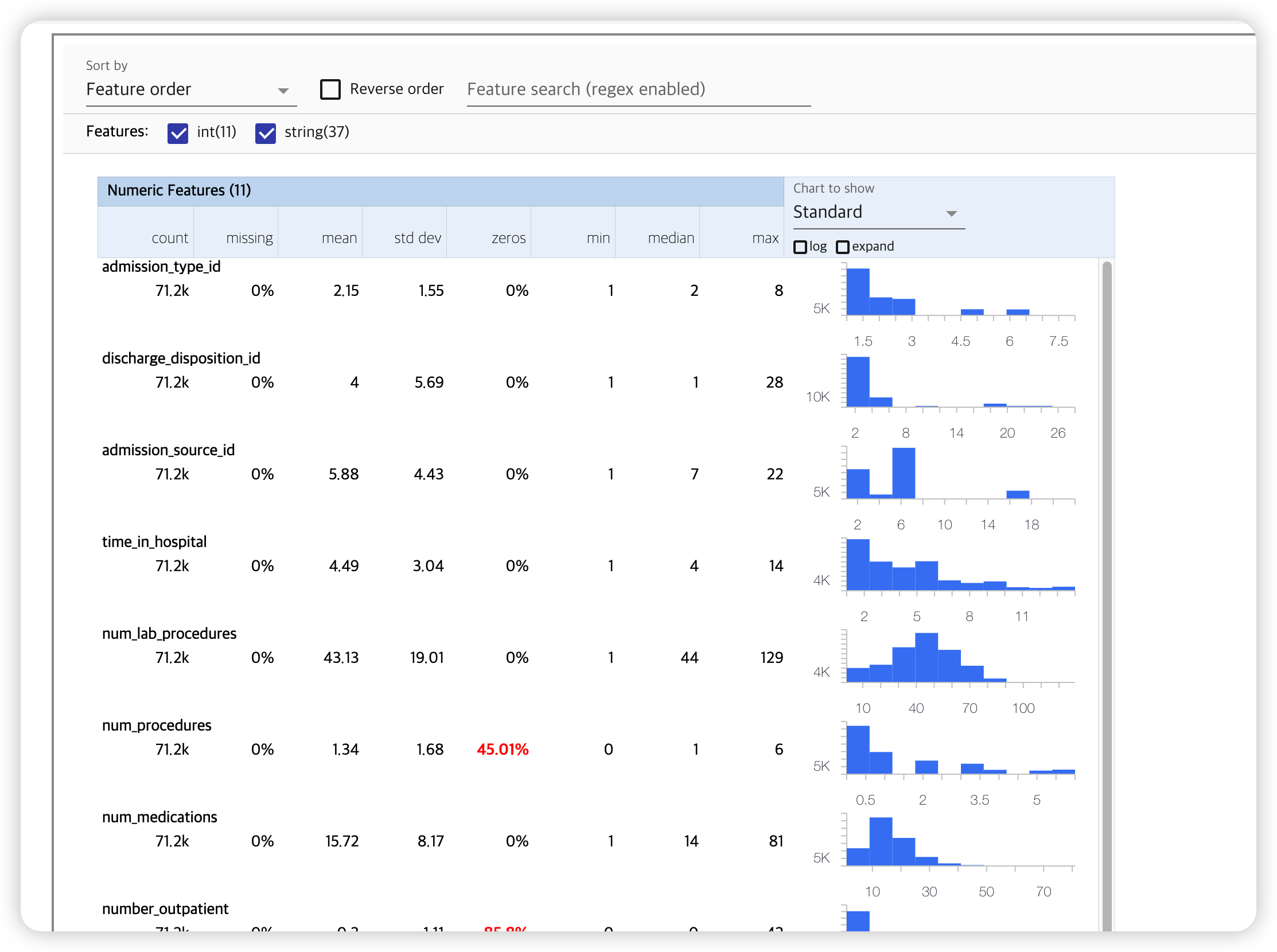
4. Infer a data schema
schema : 데이터의 특성을 정의함
- error 탐지에 사용될 수 있음
schema = tfdv.infer_schema(train_stats)
tfdv.display_schema(schema)
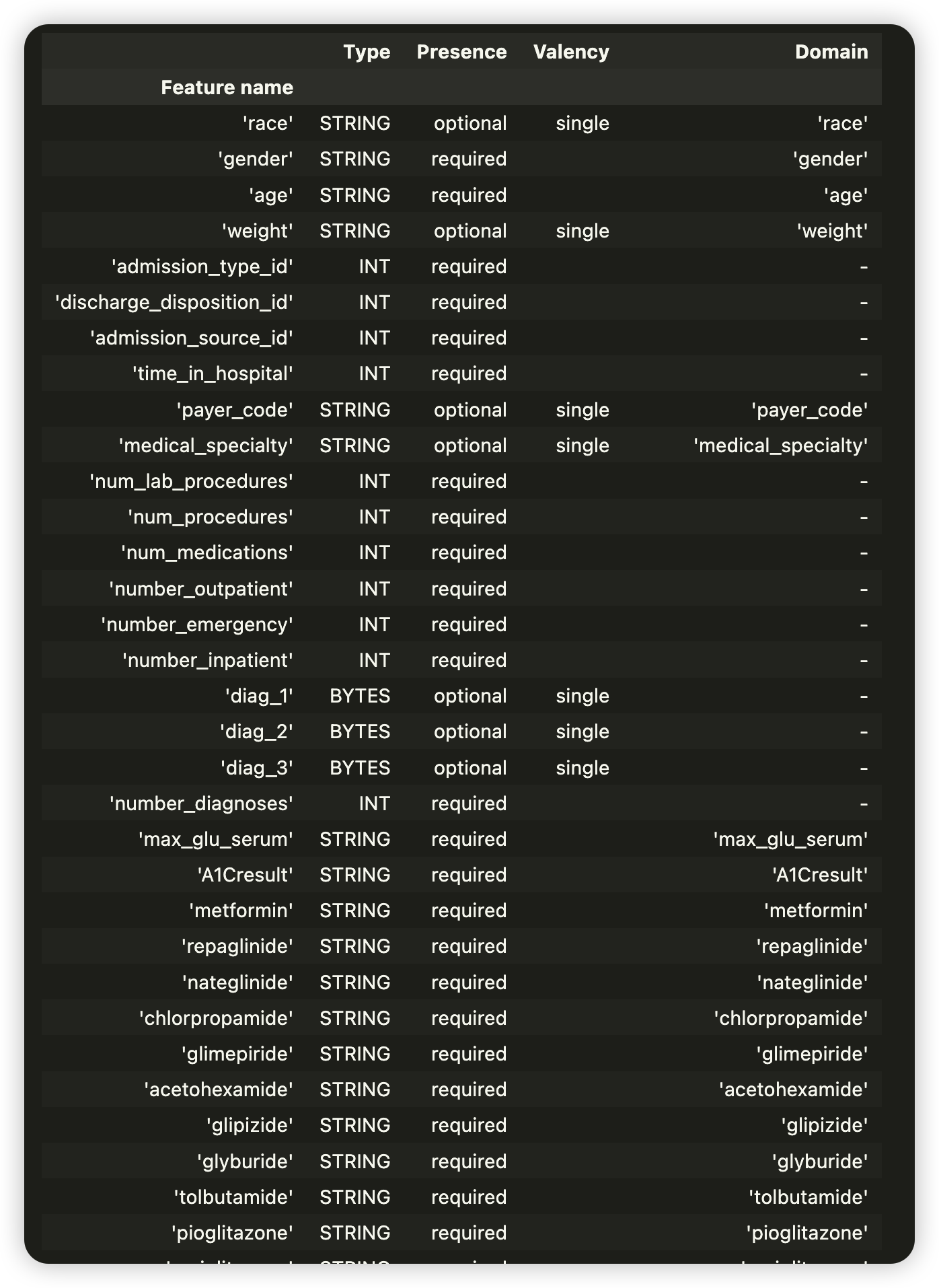
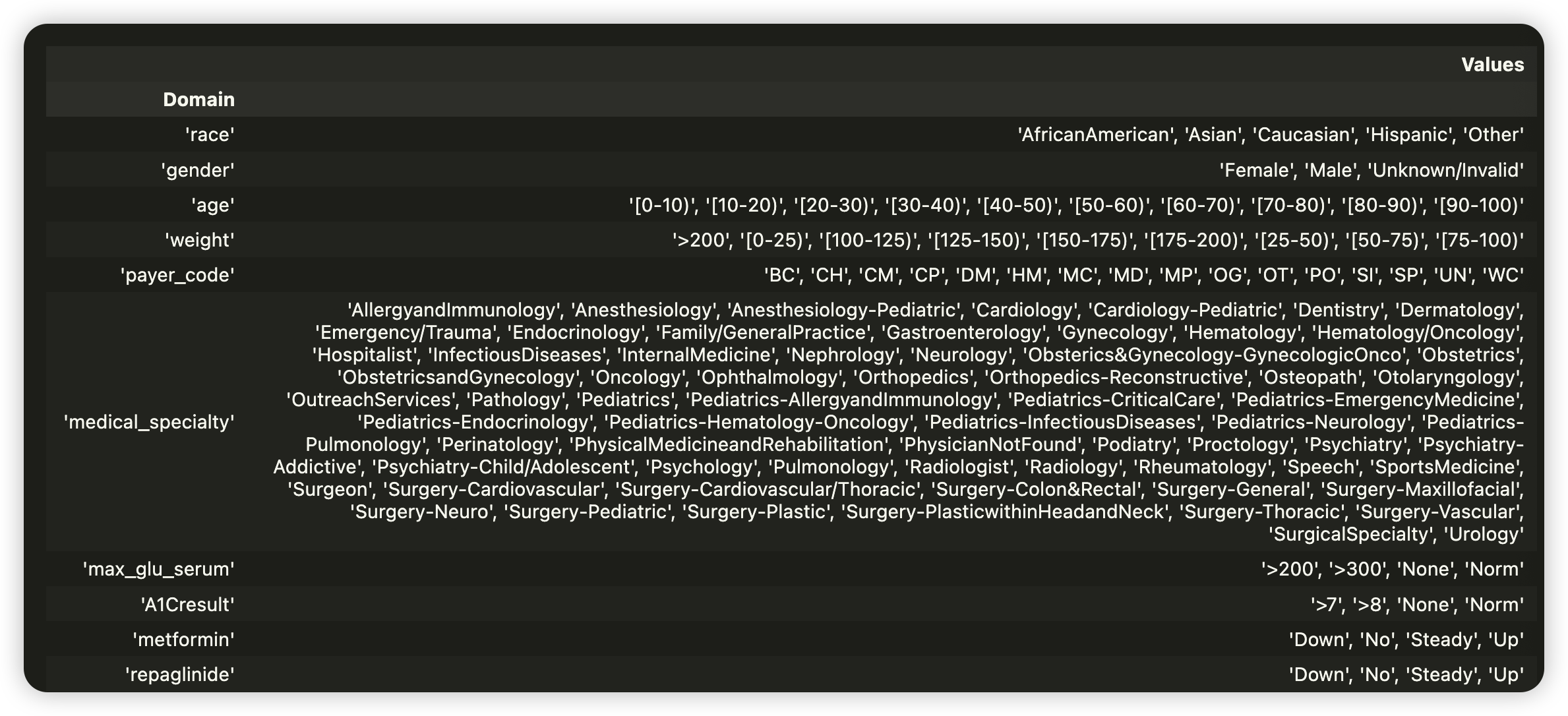
TRAINING DATASET만을 대상으로 data schema를 infer 한다
- evaluation & serving dataset을 대상으로 각각의 statistics를 계산한 뒤, 지금 이 schema와의 비교를 통해 anomaly, drift, skew등을 발견한다
len(schema.feature) # schema 내 feature의 개수 48
list(schema.feature)[1].domain # 2번째 feature
5. Calculate, Visualize, Fix Evaluation Anomalies
(1) Compare Training & Evaluation Statistics
- statistics 만들기
- 이전과 동일, but 이번에는 evaluation data 넣어주기
eval_stats = tfdv.generate_statistics_from_dataframe(eval_df, stats_options=stats_options)
- visualize
지정해야할 파라미터들
- lhs_statistics : (좌) stat
- rhs_statistics : (우) stat
- lhs_name : (좌) name
- rhs_name : (우) name
tfdv.visualize_statistics(lhs_statistics=eval_stats,
rhs_statistics=train_stats,
lhs_name='EVAL_DATASET',
rhs_name='TRAIN_DATASET')
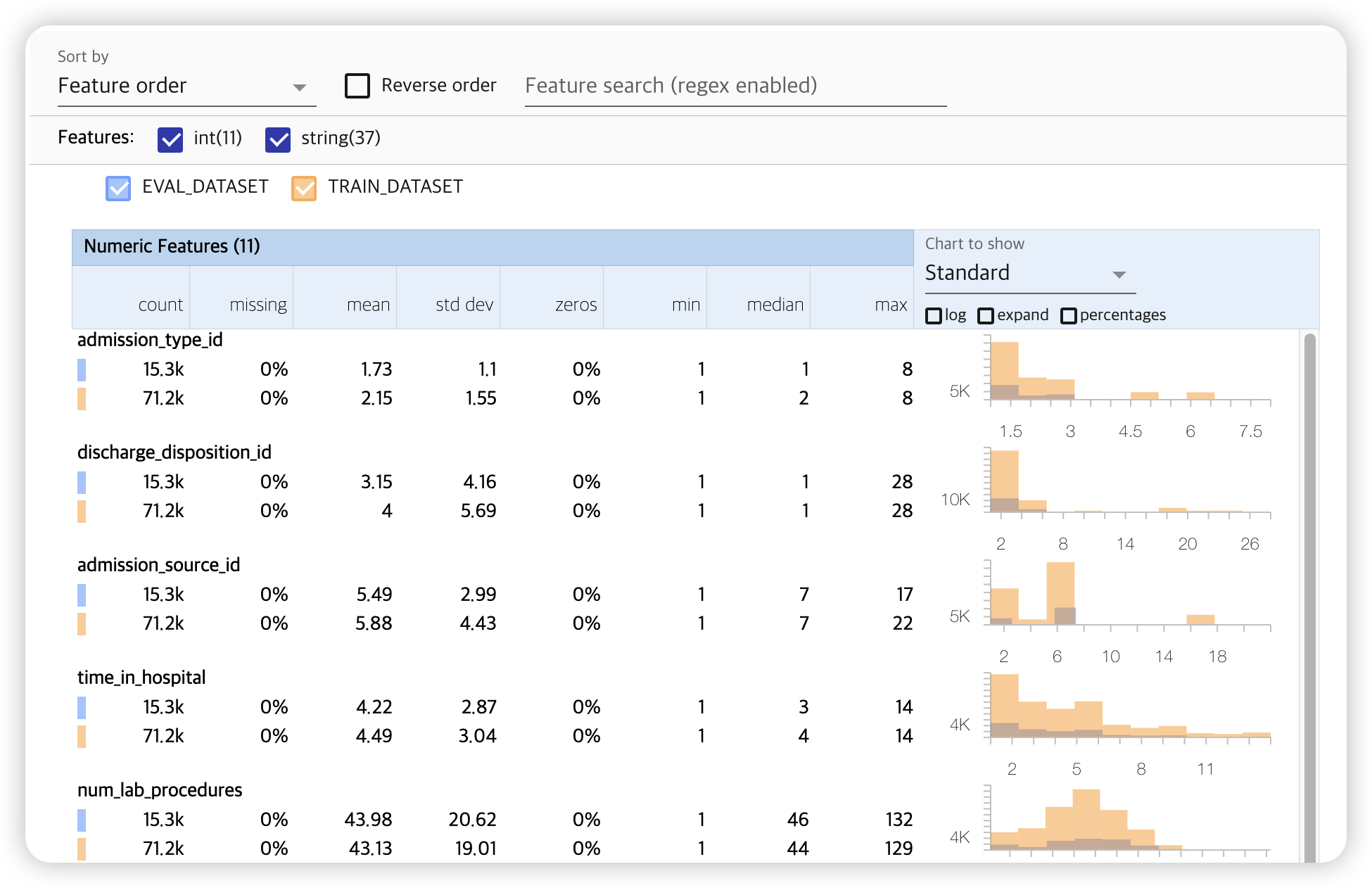
(2) Detect Anomalies
위의 시각화 dashboard를 확인해보면, 특정한 “범주형 변수 glimepiride-pioglitazone”이
Training data에 1개의 unique 값과, Evaluation data에 2개 있음을 확인할 수 있다.
train_df["glimepiride-pioglitazone"].describe()
count 71236
unique 1
top No
freq 71236
Name: glimepiride-pioglitazone, dtype: object
eval_df["glimepiride-pioglitazone"].describe()
count 15265
unique 2
top No
freq 15264
Name: glimepiride-pioglitazone, dtype: object
이걸 일일히 다 확인하기가,,,,,
TFDV function을 사용하여 확인하자!
tfdv.validate_statistics()- 인자 1) DatasetFeatureStatisticsList
- 인자 2) Schema
tfdv.display_anomalies()- 인자 ) anomalies
def calculate_and_display_anomalies(statistics, schema):
anomalies = tfdv.validate_statistics(statistics, schema)
tfdv.display_anomalies(anomalies)
확인 해본 결과, 2개의 변수에 anomaly가 있음을 확인할 수 있다.
- [ ‘glimepiride-pioglitazone’ 변수 ]
- schema ( TRAIN only )에는 없는 “Steady”라는 값이 evaluation에서 발견
- [ ‘medical_specialty’ 변수 ]
- schema ( TRAIN only )에는 없는 “Neurophysiology”라는 값이 evaluation에서 발견
calculate_and_display_anomalies(eval_stats, schema=schema)

(3) Fix evaluation anomalies in the schema
위에서, evaluation에만 발견된 값을 (train) schema에 넣어줌으로써 문제 해결!
domain.value.append(“feature_value”)
glimepiride_pioglitazone_domain = tfdv.get_domain(schema, 'glimepiride-pioglitazone')
glimepiride_pioglitazone_domain.value.append('Steady')
medical_specialty_domain = tfdv.get_domain(schema, 'medical_specialty')
medical_specialty_domain.value.append('Neurophysiology')
더 이상의 anomaly 가 발견되지 않는다 :)
calculate_and_display_anomalies(eval_stats, schema=schema)
6. Schema Environments
( 일반적으로 ) pipeline 내에 있는 모든 데이터셋들은 동일한 schema를 가져야 한다.
하지만, 예외가 있는데….
- label column : 이건 serving dataset에는 없다!
check anomalies in serving set
options = tfdv.StatsOptions(schema=schema,
infer_type_from_schema=True,
feature_allowlist=approved_cols)
이번엔, serving data를 넣어준다.
readimitted칼럼은, serving dataset을 위해 drop됨을 알 수 있다.
serving_stats = tfdv.generate_statistics_from_dataframe(serving_df,
stats_options=options)
calculate_and_display_anomalies(serving_stats, schema=schema)

Anomaly 기준 바꾸기
- Get the feature and relax to match 90% of the domain
payer_code = tfdv.get_feature(schema, 'payer_code')
payer_code.distribution_constraints.min_domain_mass = 0.9
medical_specialty = tfdv.get_feature(schema, 'medical_specialty')
medical_specialty.distribution_constraints.min_domain_mass = 0.9
calculate_and_display_anomalies(serving_stats, schema=schema)
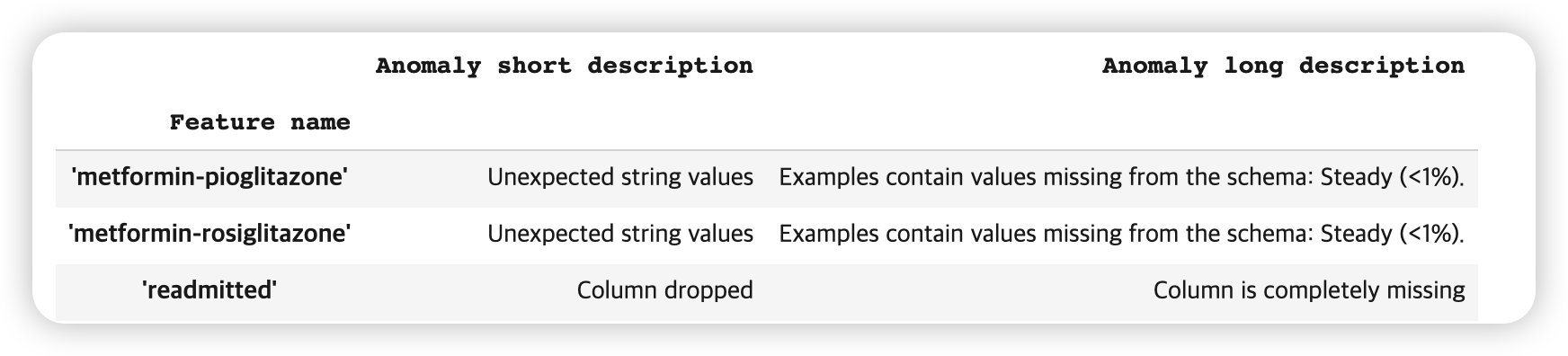
(1) Modify Domains
여러 변수에서, 가질 수 있는 값이 총 4종류 (down, no, steady, up)이 있는 것을 알 수있다.
하지만, 많은 train df내의 변수들에는, 이 4종류가 전부 없는 경우가 있다. ( 아래 사진 확인 )

따라서, 이러한 4개 미만의 값들을 가진 변수들의 도메인을 전부 변경해주자! ( overwrite )
( 4개의 종류를 모두 가지고 있는 변수 중 하나인 metformin 을 사용 )
def modify_domain_of_features(features_list, schema, to_domain_name):
for feature in features_list:
tfdv.set_domain(schema, feature, to_domain_name)
return schema
domain_change_features = ['repaglinide', 'nateglinide', 'chlorpropamide', 'glimepiride',
'acetohexamide', 'glipizide', 'glyburide', 'tolbutamide', 'pioglitazone', 'rosiglitazone', 'acarbose', 'miglitol', 'troglitazone', 'tolazamide', 'examide', 'citoglipton', 'insulin', 'glyburide-metformin', 'glipizide-metformin', 'glimepiride-pioglitazone', 'metformin-rosiglitazone', 'metformin-pioglitazone']
schema = modify_domain_of_features(domain_change_features, schema, 'metformin')
tfdv.display_schema(schema)
마지막으로 확인해보자.
calculate_and_display_anomalies(serving_stats, schema=schema)
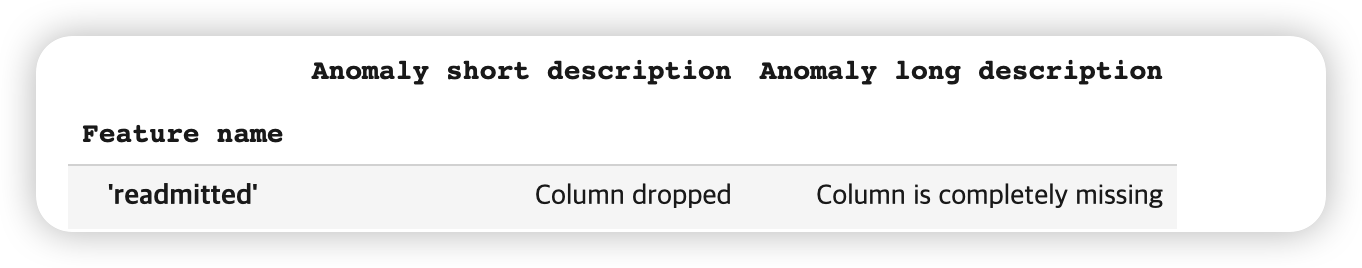
serving set에 없어야할 readmitted 칼럼만이 anomaly로 탐지되고, 나머지는 잘 해결된 것을 알 수 있다 :)
하지만…사실 이 또한 anomaly라고 볼 수 없다 ( 당연한거니까…! )
따라서, 이러한 오류(?) anomaly 문구(?)를 뜨지 않게 해보자.
schema.default_environment.append('TRAINING')
schema.default_environment.append('SERVING')
tfdv.get_feature(schema, 'readmitted').not_in_environment.append('SERVING')
serving_anomalies_with_env = tfdv.validate_statistics(serving_stats, schema, environment='SERVING')
tfdv.display_anomalies(serving_anomalies_with_env)
# no anomalies found!
7. Checking Data Drift & Skew
지금까지는 data validation 과정을 위해 anomaly detection을 했다.
하지만 이게 전부가 아니다. 우리는 data drift & data skew 또한 확인해야 한다~
특정 변수 ( diabetesMed )를 대상으로 skew 확인하기
특정 변수 ( payer_code )를 대상으로 drift 확인하기
- 기준 : L-infinity distance 0.3 ( 도메인 지식 필요 )
diabetes_med = tfdv.get_feature(schema, 'diabetesMed')
diabetes_med.skew_comparator.infinity_norm.threshold = 0.03
payer_code = tfdv.get_feature(schema, 'payer_code')
payer_code.drift_comparator.infinity_norm.threshold = 0.03
skew_drift_anomalies = tfdv.validate_statistics(train_stats, schema,
previous_statistics=eval_stats,
serving_statistics=serving_stats)
tfdv.display_anomalies(skew_drift_anomalies)

8. Display Stats for Data Slices
데이터를 slice로 나눈 뒤 ( ex. 특정 변수의 값을 기준으로 ),
이에 대한 분석을 따로 진행할 수도 있다.
# dataset들이 각각의 요소로 들어가 있는 "dataset_list"
def split_datasets(dataset_list):
datasets = []
for dataset in dataset_list.datasets:
proto_list = DatasetFeatureStatisticsList()
proto_list.datasets.extend([dataset])
datasets.append(proto_list)
return datasets
# 시각화
def display_stats_at_index(index, datasets):
if index < len(datasets):
print(datasets[index].datasets[0].name)
tfdv.visualize_statistics(datasets[index])
def sliced_stats_for_slice_fn(slice_fn, approved_cols, dataframe, schema):
# (1) slice할 옵션 ( ex. 나누는 기준 )
slice_stats_options = tfdv.StatsOptions(schema=schema,
slice_functions=[slice_fn],
infer_type_from_schema=True,
feature_allowlist=approved_cols)
# (2) df를 csv로 바꾸기
# ( slice function은, `tfdv.generate_statistics_from_csv`에서만 작동하므로 )
CSV_PATH = 'slice_sample.csv'
dataframe.to_csv(CSV_PATH)
# (3) 나누어진 dataset 바탕으로 statistics 계산
sliced_stats = tfdv.generate_statistics_from_csv(CSV_PATH, stats_options=slice_stats_options)
# (4) DatasetFeatureStatisticsList() 형태로 변환 후 반환
slice_info_datasets = split_datasets(sliced_stats)
return slice_info_datasets
# (1) 나눌 기준 설정
slice_fn = slicing_util.get_feature_value_slicer(features={'medical_specialty': None})
# (2) 나눠진 데이터셋 ( 형태 : DatasetFeatureStatisticsList를 각각의 요소로 가지는 list )
slice_datasets = sliced_stats_for_slice_fn(slice_fn, approved_cols, dataframe=train_df, schema=schema)
medical_specialty 변수에는, 총 68종류의 값들이 있다.
이 각각을 조건으로 slicing하여, 필터링할 수 있다.
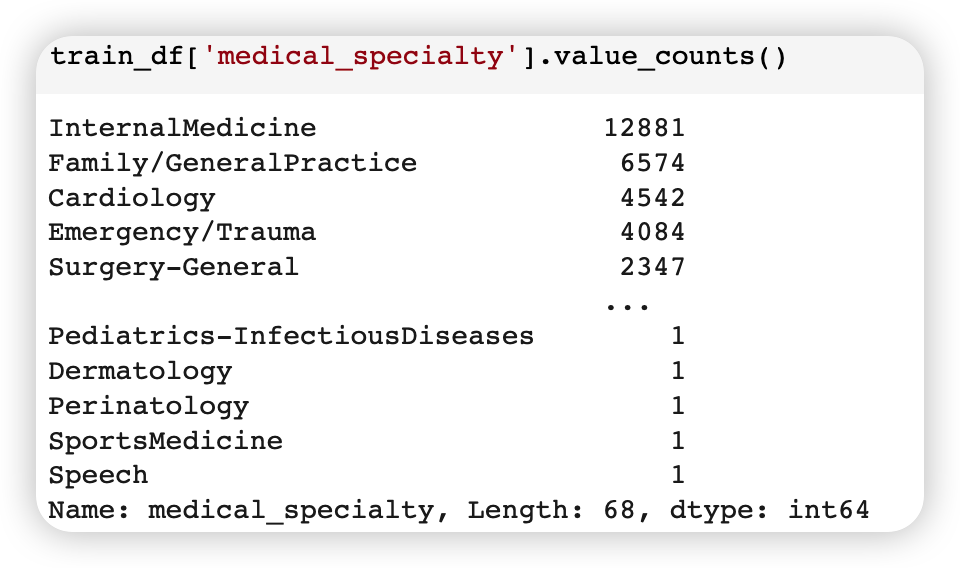
- ex) 10번째 :
medical_specialty_Gastroenterology- medical_specialty == ‘Gastroenterology’ 조건
display_stats_at_index(10, slice_datasets)
9. Freeze the schema
이제, 위처럼 만든 schema를 “frozen”된 상태로 저장할 수 있다.
이는, 새롭게 들어노느 데이터에 대해 validation을 진행할 때 사용할 수 있다!
OUTPUT_DIR = "output"
file_io.recursive_create_dir(OUTPUT_DIR)
schema_file = os.path.join(OUTPUT_DIR, 'schema.pbtxt')
tfdv.write_schema_text(schema, schema_file)
